两篇Nature “比翼齐飞”,中国科学家揭示生长素极性运输的分子机制
植物激素新受体发现

图1.中科大与浙大研究团队以“背靠背”的形式在Nature上发表了两篇关于生长素转运的研究论文
中国科学技术大学研究团队与浙江大学研究团队在这两篇研究论文中(图1),同时报道了植物中生长素极性转运蛋白PIN以及PIN与生长素IAA结合、PIN与除草剂NPA结合的三个高分辨率结构,并结合Biacore分子互作技术阐明了膜蛋白PIN与生长素IAA(分子量175 Da)和除草剂NPA的识别和转运机制,为剖析植物生长素运输调控以及针对PIN蛋白的农业用除草剂和植物生长调节剂的设计开发奠定了重要基础。
首先,浙江大学研究团队通过体外放射性IAA(生长素)转运试验,证明了PIN3可以运输 IAA,加入抑制剂NPA后,NPA有效抑制了IAA的流出,从而证实了IAA流出是由PIN3 控制的。进一步,科研人员解析了PIN3、PIN3-IAA 和PIN3-NPA的结构,并发现他们结构十分类似。PIN3以同源二聚体的形式存在,具有垂直于膜的C2对称性;并且,IAA和NPA与PIN的结合方式相似。
为验证IAA在蛋白PIN3上的结合位点与关键氨基酸,科研人员通过Biacore实验发现,膜蛋白PIN3与IAA直接结合,亲和力KD为160.4 μM(图2);对PIN3蛋白上关键氨基酸进行单点突变(V51A, N112A, L114A, V137A, Q140A, Y145A, I600A和V601A)后,其与IAA的亲和力显著降低,其中Y145A突变后亲和力降低最多,为402.2 μM(图3),实验结果表明,这些氨基酸位点对于IAA与PIN3的结合均很重要,从而确定了IAA在PIN3上结合的关键氨基酸,与结构的数据高度一致。
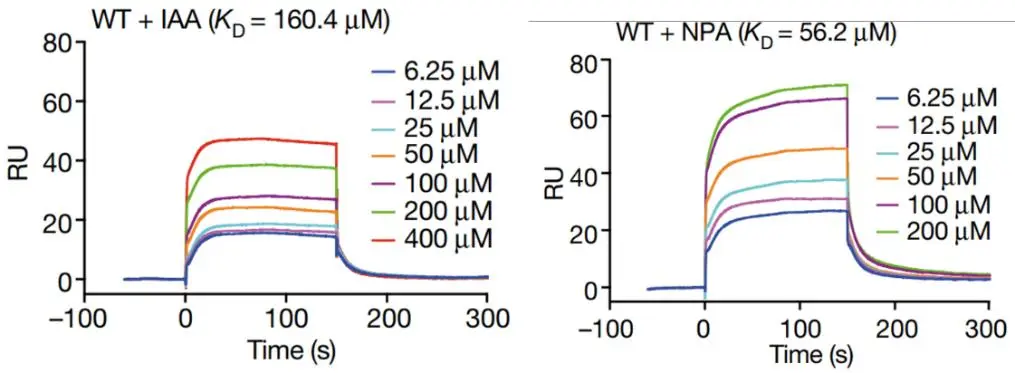
图2.Biacore实验检测,野生型PIN3(WT)与生长素IAA及其与除草剂NPA的亲和力
为进一步验证除草剂NPA的结合位点是否与IAA结合位点相同,以及NPA抑制IAA的分子机制,科研人员通过Biacore实验发现,膜蛋白PIN3与NPA直接结合,亲和力KD为56.2 μM,强于IAA与PIN3的亲和力(160.4 μM)(图2);对PIN3蛋白上IAA结合的关键氨基酸位点进行单点突变(V51A, N112A, L114A, V137A, Q140A, Y145A, I600A和V601A)后,结果同样显示,突变后与NPA的亲和力显著降低(图3),表明 NPA与PIN3的结合位点与IAA相同。因此,NPA通过直接结合PIN3蛋白上的IAA结合位点,以较高的亲和力抑制PIN3的IAA转运活性。最终,阐明了PIN3与生长素IAA和除草剂NPA的识别和转运机制。
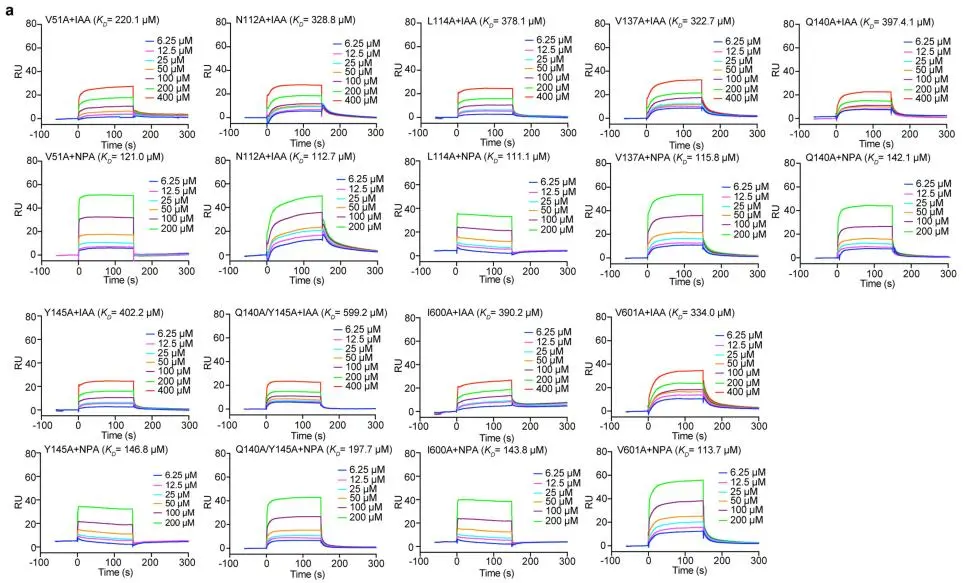
图3.Biacore实验检测,各突变型PIN3与生长素IAA及其与除草剂NPA的亲和力
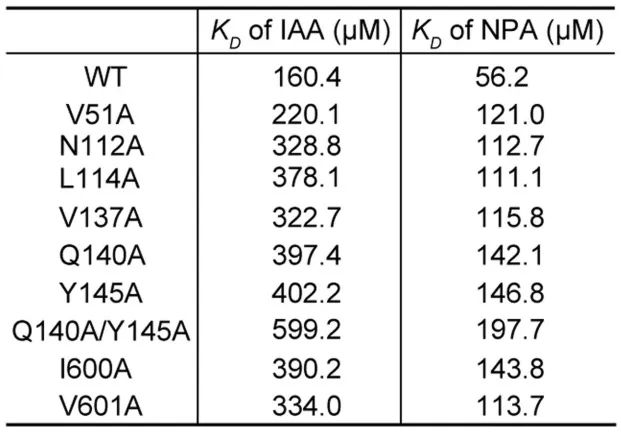
表1.Biacore实验检测,各突变型与野生型PIN3与生长素IAA及其与除草剂NPA的亲和力
正所谓“一蛋双黄,好事成双” 。中国科学技术大学研究团队同样地解析了PIN蛋白家族另一个成员PIN1、PIN1-IAA 和PIN1-NPA的结构。并且,他们通过Biacore实验还发现, PIN1也能与生长素IAA直接结合,亲和力KD为186 μM(图4),与PIN3与IAA的亲和力(KD为160.4 μM,图2)相当;同时,他们还检测了PIN1与其他天然生长素(IBA、IPA、4-Cl-IAA)均能结合(图4)。
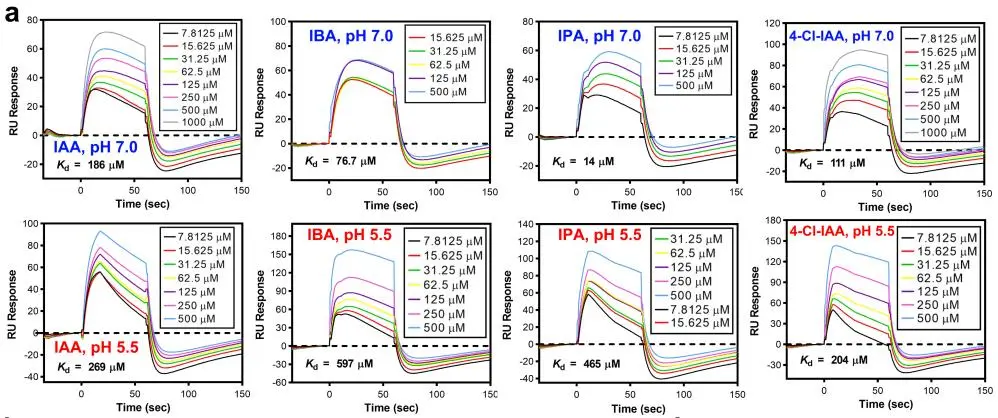
图4.Biacore实验检测, PIN1与四种天然生长素IAA、IBA、IPA、4-Cl-IAA的亲和力
- 小分子IAA分子量较低(175 Da),结合产生的信号不会太高,这对检测设备的灵敏度提出了更高的要求,Biacore以其卓越的灵敏度,对于小分子的检测无分子量下限,具有高达pg级别的超高灵敏度0.03 RU (RMS),能够确保低信号的结合也能精确检测;
- PIN是膜蛋白,结构不稳定、表达量低,较难纯化;并且纯化得到的蛋白也面临着极易失活等问题。因此,找到一种既能保证PIN蛋白活性状态,又能对其开展理化性质表征的检测技术尤为关键。Biacore分子互作系统即是采用了一种持续流检测技术,时刻保证PIN蛋白处于溶液状态,并且无需对分子进行标记处理,减少对蛋白空间构象的影响,为膜蛋白的检测保驾护航。
同一期的两篇Nature文章,出自国内两个顶级科研单位。但他们却不约而同的同时选择使用了 Biacore技术,这怎么就那么巧呢?其实,看似巧合,实则必然。Biacore作为分子互作检测的“金标准”,以其卓越的性能,为不同领域的科学研究提供了大量精准的数据,已经助力超过五万篇文献的发表,成为了发表高分文章的必备工具。
参考文献:
Yang, Zhisen et al. “Structural insights into auxin recognition and efflux by Arabidopsis PIN1.” Nature vol. 609,7927 (2022): 611-615.
Su, Nannan et al. “Structures and mechanisms of the Arabidopsis auxin transporter PIN3.” Nature vol. 609,7927 (2022): 616-621.

 收藏
收藏 询价
询价