一年连发6篇高分文章 | 快来瞧瞧他们用了啥?!
延期了一年的奥运会正在hot的东京如火如荼的举行,来自世界各地的奥运健儿们在奥运赛场上争分夺秒、争金夺银。而同样正在攻克科研难题的你,是否也期待着夺得属于自己的“金牌”?
俗话说“工欲善其事,必先利其器”。高分文章的发表,绝对离不开优秀的实验设备和技术手段。今天为大家介绍一款基础科研中的“夺金”利器——Biacore,它在一年左右的时间里,就帮助清华大学张林琦教授团队发表了2篇《Nature》、1篇《Immunity》、2篇《Nature Communications》与1篇《Cell research》。
- 在基础科研中,你会不会因为需要涉猎的检测技术太多而烦恼?
- 因不同检测结果匹配不上而怀疑人生?
- 因频频出现的假阴(阳)性结果而多次返工?
2021年7月9日,清华大学医学院、结构生物学高精尖创新中心、国家感染性疾病临床医学研究中心、香港大学医学院与中国食品药品检定研究院等单位在《Nature Communications》杂志上发表了《Potent and protective IGHV-53/3-66 public antibodies and their shared escape mutant on the spike of SARS-CoV-2》文章,揭示了新冠病毒突变对不同患者体内已经产生的中和抗体造成了严峻挑战,为新一代新冠广谱疫苗设计提供科学基础。这也是清华大学张林琦教授团队在新冠抗体研究中的又一杰出成果。
1. 病毒入侵机制解读
2020年3月,课题组在《Nature》上发表文章《Structure of the SARS-CoV-2 spike receptor-binding domain bound to the ACE2 receptor》,准确定位了新冠病毒RBD和受体ACE2的相互作用位点,用Biacore直接表征了病毒RBD与ACE2受体相互作用的过程(图1),揭示了ACE2特异性介导新冠病毒细胞侵染的结构基础,为治疗性抗体药物开发以及疫苗的设计奠定了坚实基础。
![covid19_2_pic1 图1 SARS-CoV-2 RBD、SARS-CoV RBD与ACE2的动力学表征 [1]](https://cytivalearning.uin88.com/wp-content/uploads/2025/09/covid19_2_pic1.webp)
图1 SARS-CoV-2 RBD、SARS-CoV RBD与ACE2的动力学表征 [1]
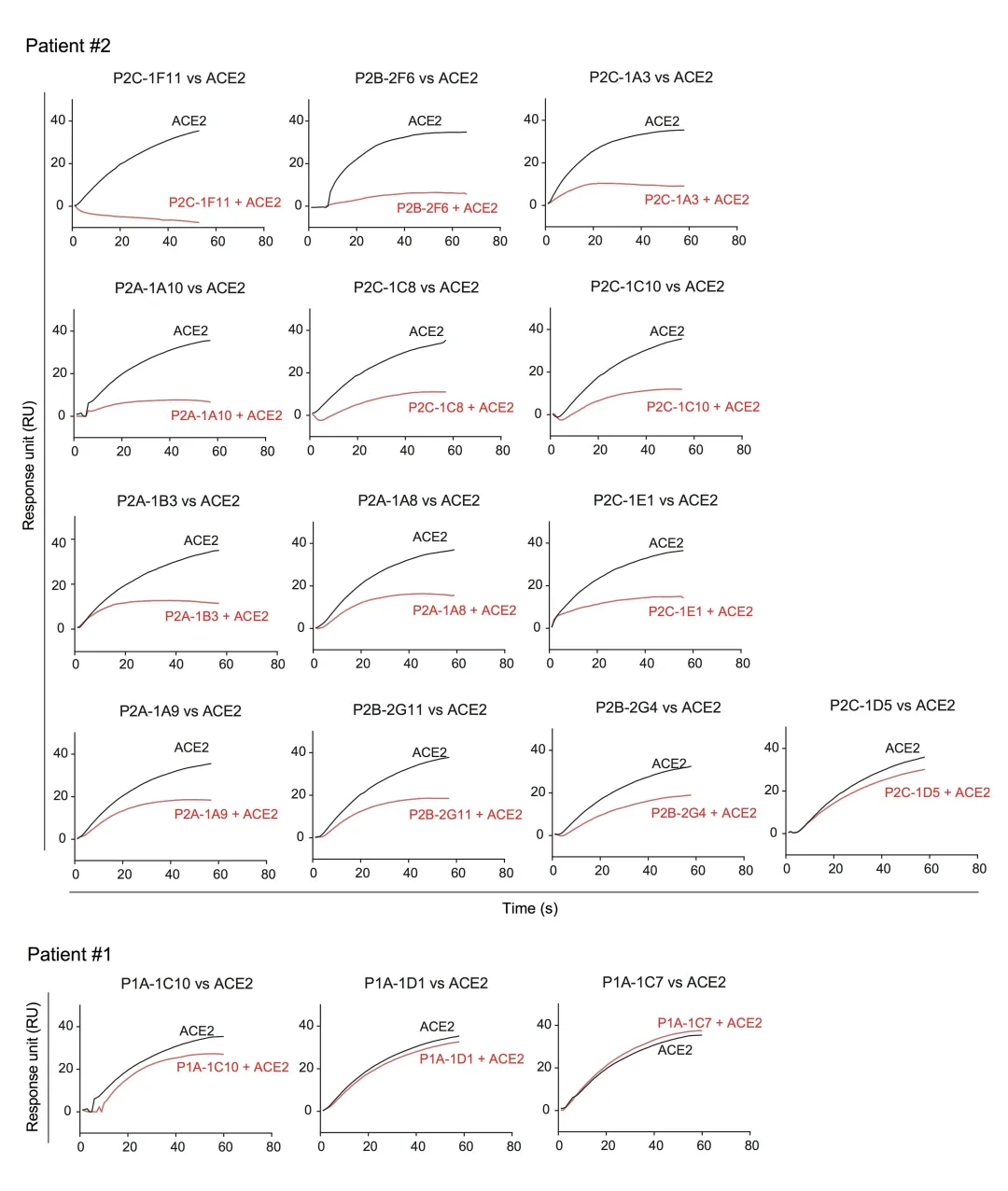
2020年5月,课题组在《Nature》上发表文章《Human neutralizing antibodies elicited by SARS-CoV-2 infection》。研究人员从8例SARS-CoV-2感染者的单个B淋巴细胞中分离并鉴定出了206个特异性靶向RBD的单克隆抗体,发现这206个单克隆抗体的中和活性与其竞争ACE2结合RBD的能力密切相关。在此篇文章中,研究人员借助Biacore巧妙的设计了表位鉴定(epitope binning)和竞争筛选实验(competition screen)(图2),助力找到一系列高活性中和抗体(P2C-1F11、P2B-2F6、P2C-1A3等)。
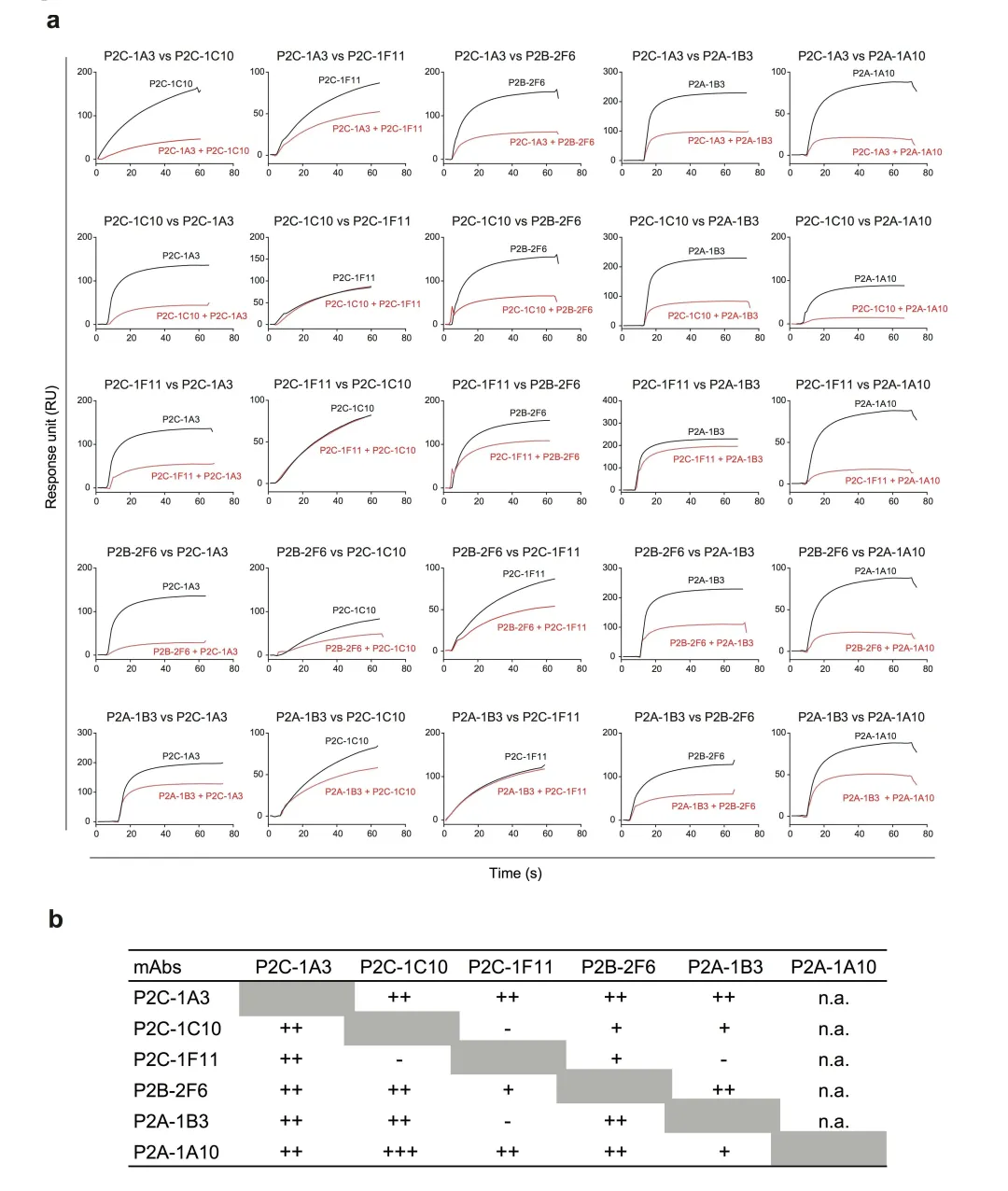
2021年1月,为探究从新冠病人体内分离出的高活性中和抗体(P2C-1F11、P2B-2F6、P2C-1A3)的结构信息及作用机制,研究人员展开了免疫复合物的晶体培养-结构解析及动力学表征工作,并在《Nature Communications》上发表了文章《Antibody neutralization of SARS-CoV-2 through ACE2 receptor mimicry》。
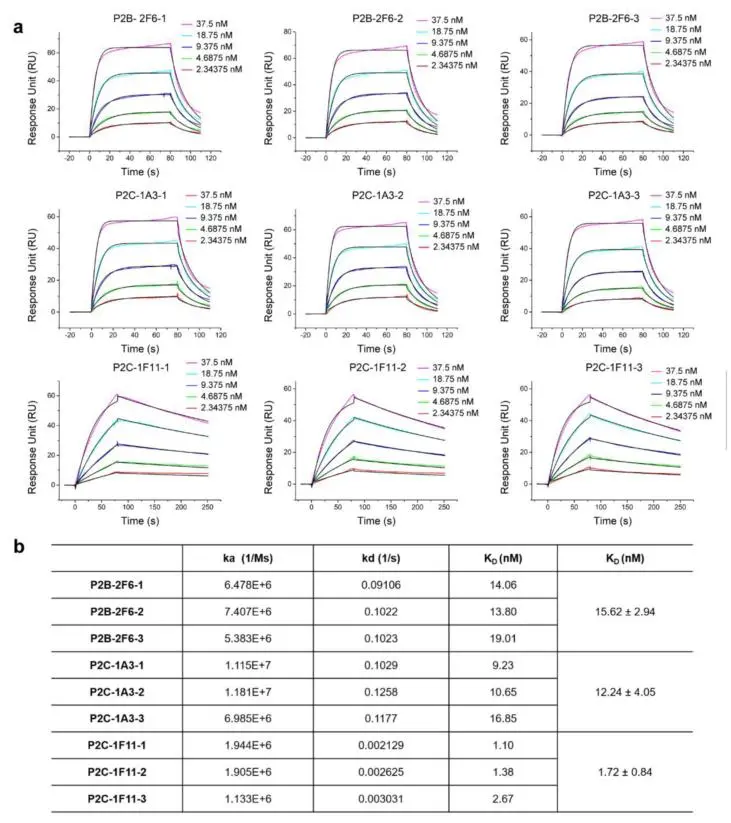
从Biacore上得到的动力学检测数据表明P2C-1F11与RBD的亲和力KD是三个单抗中最强的(1.72±0.84 nM)。与Biacore得到的结果一致,通过比较不同免疫复合物的surface图,可以看到P2C-1F11与SARS-CoV-2 RBD的表面印迹面积(binding interface)也是最大的(995 Å2)(图3)。
文章中不同检测技术均可引向同一结论:P2C-1F11可诱导Spike蛋白S1亚基发生脱落,从而阻断病毒入胞。实验结果表明,3株单抗与RBD的亲和力均在nM水平,而Biacore的灵敏度及数据准确度为科研工作者保驾护航,将不同抗体间极小的差异也能精确地表征。
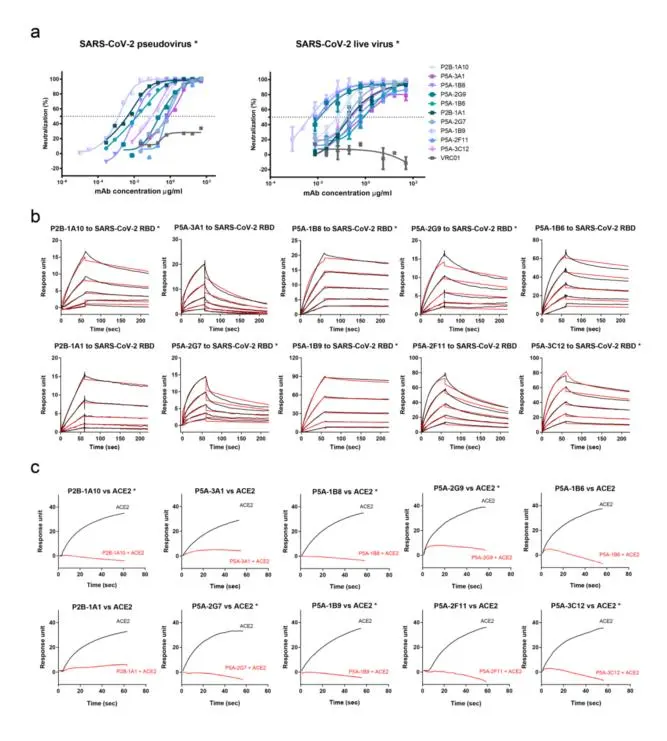
针对从8例新冠病毒感染者的B细胞中分离并鉴定出了206株特异性靶向RBD的单克隆抗体,研究团队还解析了10株靶向SARS-CoV-2 RBD的高效中和抗体与Spike蛋白的复合物结构,深入研究了IgG形式二价抗体结合S蛋白的结构基础与其诱导S1蛋白脱落(Shedding)的可能分子机制 。研究课题组使用Biacore完成了相关抗体与RBD的动力学表征及ACE2的竞争分析实验(图4)。相关研究成果于2021年3月发表在了在《Cell research》上:《Structural basis for bivalent binding and inhibition of SARS-CoV-2 infection by human potent neutralizing antibodies》。
2021年6月,研究团队在《Immunity》上发表文章《Analysis of SARS-CoV-2 variant mutations reveals neutralization escape mechanisms and the ability to use ACE2 receptors from additional species》。研究团队通过假病毒中和实验、解析免疫复合物晶体结构、借助Biacore设计并完成动力学表征实验等,从细胞水平、原子水平、分子水平逐一解析了新冠病毒突变株逃逸抗体中和的分子机制。
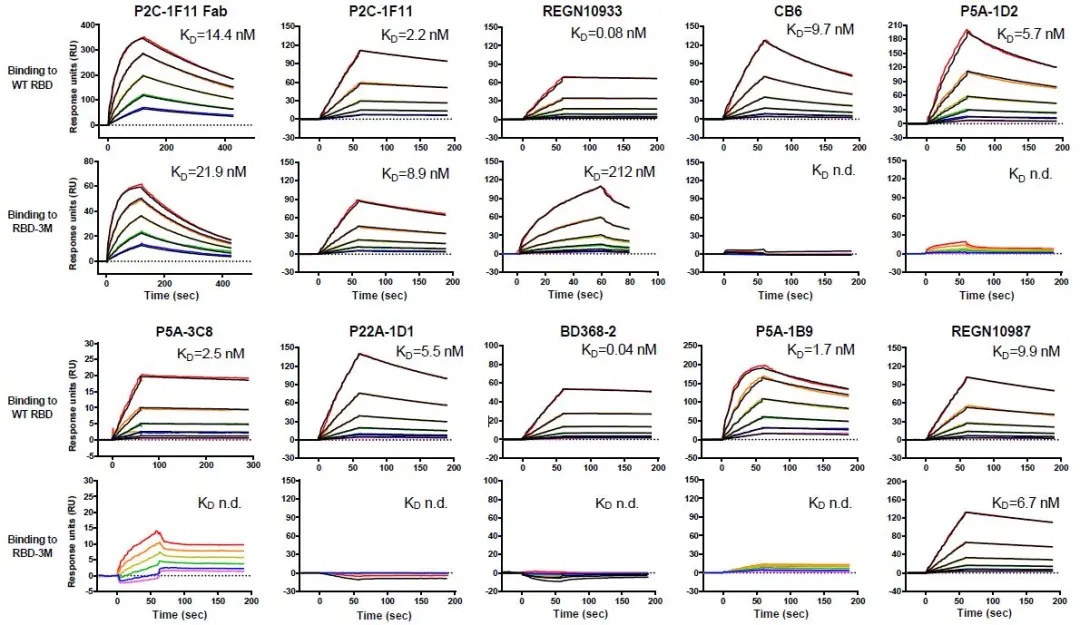
2021年7月,研究团队在《Nature Communications》上发表文章《Potent and protective IGHV-53/3-66 public antibodies and their shared escape mutant on the spike of SARS-CoV-2》。文中发现不同患者体内均能诱导出IGHV3-53/3-66基因谱系的抗体,被称为公共抗体(public antibody)。研究团队分析四株公共抗体与RBD复合物的结构信息,通过经典的点突变方式并借助Biacore剖析了不同公共抗体与RBD发生相互作用的关键氨基酸位点,发现该类公共抗体与RBD的结合角度、结合表位非常相似,与ACE2均有强竞争能力。该类公共抗体对新冠病毒野生株具有强效中和能力,仓鼠动物模型的保护实验也证明该类抗体对新冠病毒野生株具有非常出色的保护能力。
![covid19_2_pic8 图6 5株公共抗体与SARS-CoV-2 RBD的动力学表征[6]](https://cytivalearning.uin88.com/wp-content/uploads/2025/09/covid19_2_pic8.webp)
看到这儿,你是否已经为Biacore竖起大拇指了呢?话不多说,赶紧设计实验方案做实验吧!(悄悄说一句:相关Protocol,例如竞争分析(competition screen)、抗原表位作图(epitope binning)、抗体筛选(antibody screen)等,均可从Cytiva官网下载获得哦!)
参考文献(上下滑动查看完整):

 收藏
收藏 询价
询价