全新的Poly进样模式助力复合物形成机制研究
全新的Poly进样模式
案例一
DNA错配修复复合物的形成
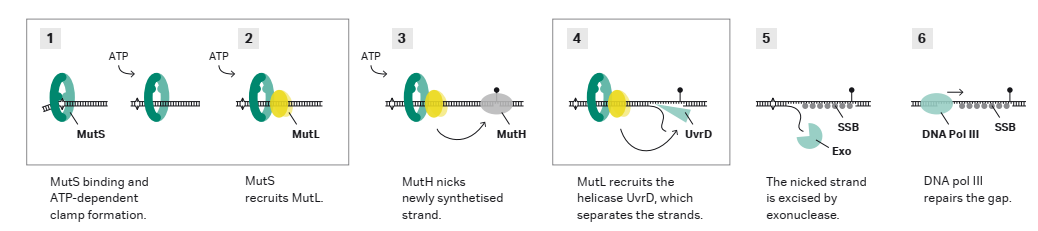
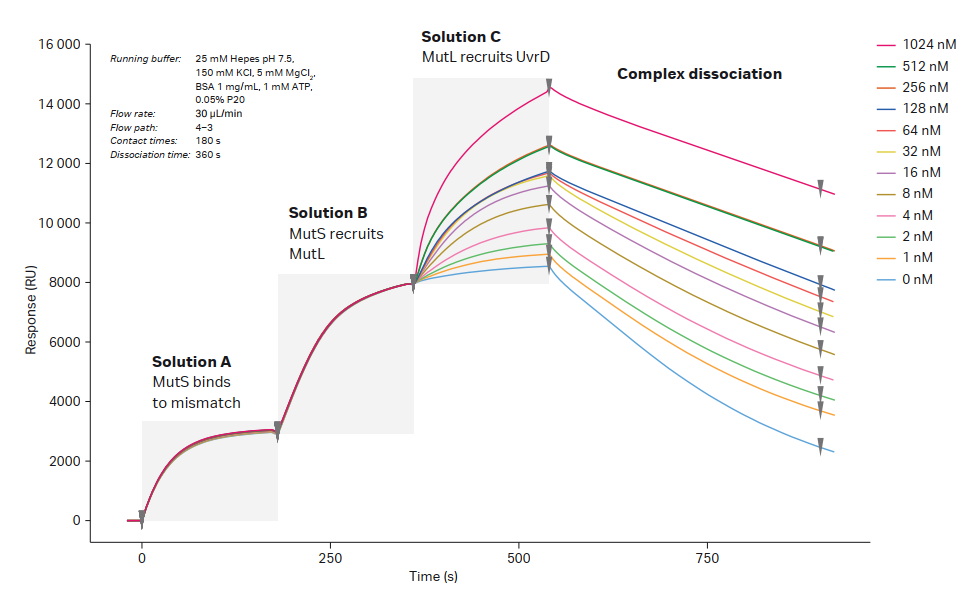
DNA错配修复是一种高度保守的生物学途径,用于检测和去除复制过程中引入的DNA错配,维持基因组稳定性。DNA错配修复复合物形成主要有三个阶段(见图2中步骤1/2/4):
- 首先MutS与DNA中错配位置结合。
- 随后MutS募集MutL。
- 最后MutL招募UvrD结合,从而进行DNA修复。为验证DNA错配修复复合物形成的过程,我们可以采用Poly进样模式,完美还原了组装顺序。
案例二
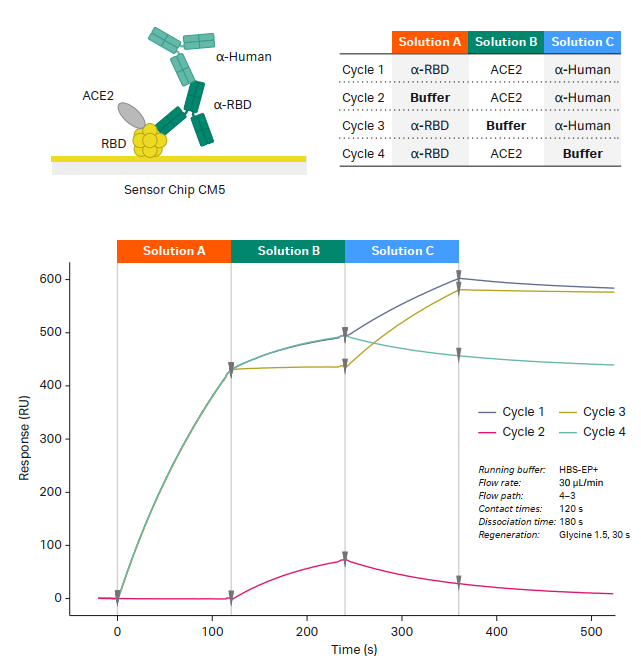
新冠相关蛋白复合物中各组分的结合依赖性
该新冠相关蛋白复合物由新冠病毒刺突蛋白受体结合域 (RBD) 、RBD受体 (ACE2) 、结合RBD的抗体CR3022 (α- RBD) 、抗人抗体 (α-Human) 组成。那么该复合物中各组分结合依赖性如何呢?首先,将RBD固定在S系列CM5芯片上。紧接着,Cycle 1:使用Poly命令顺序进样α-RBD (10 nM) 、ACE2 (750 nM) 和α-Human (10 nM) 。Cycle 2-4:每个循环中,一个组分用Buffer代替(图4)。
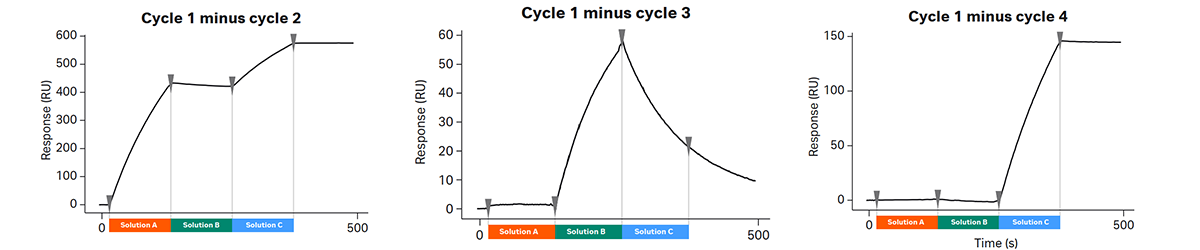
Poly进样模式总结

满足对更先进的分析工具的需求,用于表征复合物的形成

支持3~5种组分的顺序进样,最大限度地减少进样之间的解离时间

可用于分析多个分子间的结合依赖性
参考文献:
Cynthia S, Alexander F, Elin S, Linnea N, et al.Visualize your research – SPR for protein complex formation studies. 官方材料链接KZCx_Xxvx0-OzoZ3bfpzpg-pdf (cytivalifesciences.com)
Fernandez-Leiro R, Bhairosing-Kok D, Kunetsky V, et al. The selection process of licensing a DNA mismatch for repair. Nat Struct Mol Biol. 2021;28(4):373-381. doi:10.1038/s41594-021-00577-7
Yuan M, Wu NC, Zhu X, et al. A highly conserved cryptic epitope in the receptor binding domains of SARS-CoV-2 and SARS-CoV. Science. 2020;368(6491):630-633. doi:10.1126/science.abb7269

 收藏
收藏 询价
询价