从诺奖看未来:SPR助力AI推动蛋白质结构预测的革新之路!
2024年,一直以“学界桂冠”著称的诺贝尔化学奖授予了David Baker、Demis Hassabis和John M. Jumper,以表彰他们在蛋白质结构预测领域的开创性贡献,尤其是在将人工智能(AI)与计算化学相结合,推动蛋白质结构研究取得革命性进展。

通过这一突破,科学家们得以以前所未有的精度预测蛋白质的三维结构,极大地加速了生物医学研究和药物开发的进程;而诺奖的授予,也让蛋白质结构研究这一话题走入了更多人的视野之中。
设计与靶点高亲和力和特异性结合的蛋白质是计算结构生物学中一个长期的挑战,具有在治疗学、诊断学等领域的应用。为了解决这个问题,大多数蛋白质设计方法通常依赖于预先存在的支架——无论是天然蛋白质还是全新设计的蛋白质——都需要具有明确的三级结构。使用支架作为基础的优点在于,在序列设计之后,相当一部分设计可能如预期般折叠,但这限制了形状匹配的范围,特别是对于表面相对平坦、缺乏小型微蛋白可嵌入凹陷的目标。
针对现有问题,David Baker教授及其团队开发了一种能从头设计生成全新蛋白质的人工智能算法:RFdiffusion,其可以从完全随机的残基分布开始,在感兴趣的靶点的存在下,没有任何预先的支架的指导下进行。完全无约束的RFdiffusion可以生成多种多样的折叠和组装,以及包裹在延伸的螺旋肽周围的折叠蛋白质。
2024年12月5日,David Baker教授及其团队在Science上发表了“Target-conditioned diffusion generates potent TNFR superfamily antagonists and agonists”一文。
由于肿瘤坏死因子受体超家族(TNFRSF)中包括许多重要的药物靶点,但很多家族成员缺乏一些能够靶向的、具有一定疏水性位点的凹陷区域。该文章主要专注于TNFR超家族进行拮抗剂和激动剂与靶点的研究。
首先是针对TNFR1,David Baker教授及其团队首先通过RFdiffusion算法筛选了多个主链结构并预期既能够折叠成目标结构,又能跟靶标结合。并将96个设计的基因在大肠杆菌中表达。
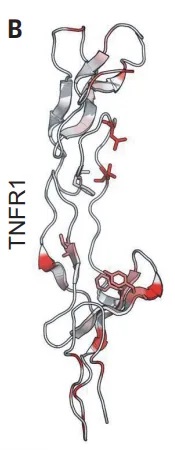
Fig1,TNFR1是一个具有挑战性的靶点
其表面平坦且只有少量表面疏水残基
接下来,David Baker教授及其团队运用Biacore验证设计蛋白与TNFR1的结合,其中蛋白TNFR1_mb1和TNFR1_mb2的KD分别为29 nM和24.5 nM。两种设计都具有高度特异性,对TNFR2没有检测到可见的结合。
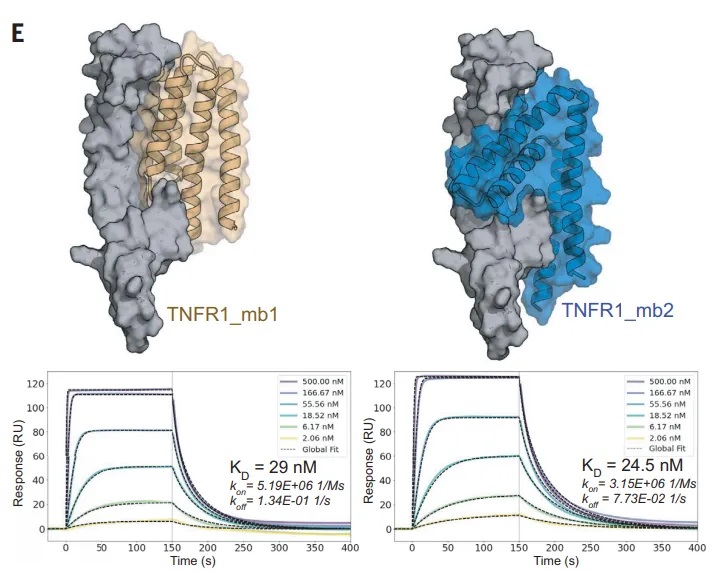
Fig.2 设计蛋白与TNFR1结合的设计模型及其SPR检测结果
接下来作者又用了部分扩散 (Partial diffusion),将主链部分加噪后又通过RFdiffusion去噪并将蛋白表达。通过Biacore检测设计蛋白与TNFR的结合,其中30%能与TNFR1结合,且将TNFR1_mb2的亲和力提高了三个数量级至=<10 pM,而TNFR1_mb3的亲和力则从微摩尔范围的弱结合提高到20 nM。部分扩散的TNFR1_mb2(TNFR1_mb2_pd1)的低皮摩尔亲和力远高于任何以前描述的单体TNFR1结合蛋白的亲和力。

Fig3. 部分扩散设计后对于亲和力的影响
鉴于部分扩散对于提高蛋白亲和力方面的成功,文章又用类似的方法对TNFR超家族的其他成员TNFR2、OX40和4-1BB进行计算设计。对于TNFR2,32%的设计具有高特异性结合,亲和力最高的设计对TNFR2的KD为198 pM,对测试的其他家族成员没有亲和力。通过TNFR1结合物开始的部分扩散预测了一个对OX40的结合物,其KD为30 nM。对于与TNFR1关系更远的4-1BB,则需要额外一轮部分扩散,但实验成功率仍然高,测试的48个设计中有22个特异性结合其目标,亲和力最高为44 nM。
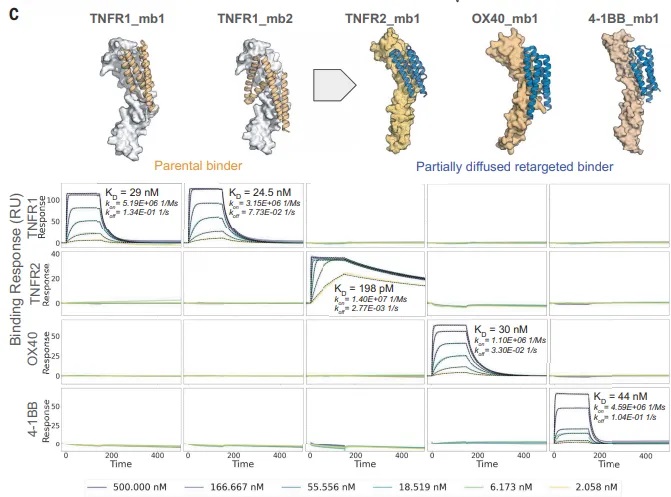
Fig4. 亲和力最强的TNFR2、OX40和4-1BB结合物的设计模型
除了此文章外,David Baker教授及其团队成功开发了多款重要的软件工具,帮助蛋白质研究中的结构预测和设计。在此基础上,Baker的团队也成功设计了多种具有特定功能的新蛋白质,在新型生物治疗药物开发、疫苗、和诊断等方面都具有重要贡献。
-
Biacore作为表面等离子共振(SPR)技术的先锋,具有高检测准确度、广泛的亲和力覆盖范围等显著特点,为AI蛋白设计提供了强大的助力。
-
Biacore特有的智能数据质量评估系统,能够以图形化显示方式评估检测结果,自动进行统计学分析,并给出相应参数,以此判断数据的可信度与准确度。
这种数据质量的保证对于AI蛋白设计的优化和验证至关重要,确保了实验结果的可靠性和可重复性。Biacore不仅在基础科研与药物研究中发挥着重要作用,还在持续为AI蛋白设计提供关键的实验数据支持,推动了蛋白质结构预测和设计的革命。

 收藏
收藏 询价
询价